На положительный исход программ вспомогательных репродуктивных технологий (ВРТ) влияет множество факторов, среди которых ключевыми являются качественный эмбрион, рецептивный эндометрий и установление «диалога» между ними [1, 2]. Однако далеко не во всех случаях перенос эмбриона хорошего качества в полость матки в соответствующей фазе менструального цикла приводит к наступлению беременности [3]. Значительный вклад в этиологию неудач имплантации и ранних репродуктивных потерь беременности вносят хромосомные аномалии эмбрионов. Известно, что более половины случаев ранних репродуктивных потерь беременности ассоциированы с анеуплоидными эмбрионами [4]. А частота образования эмбрионов с хромосомными аномалиями имеет устойчивую корреляцию с возрастом женщины. Так, если у женщин в возрасте 26–30 лет доля анеуплоидных эмбрионов составляет 20–27%, то в возрасте 45 лет 95,5% эмбрионов имеют хромосомные аномалии [5, 6]. Учитывая современную тенденцию к более позднему деторождению, образование анеуплоидных эмбрионов имеет критически важное значение, а возможность выявления эмбрионов с хромосомными аномалиями на доимплантационном этапе особенно актуальна.
Используемые в клинической практике визуальные методы оценки качества эмбрионов не отражают хромосомный статус и не исключают переноса анеуплоидного эмбриона в полость матки [7]. Около 44,5% анеуплоидных бластоцист имеет нормальное морфологическое строение [8]. Единственным на сегодняшний день методом оценки плоидности эмбрионов в клинической практике является преимплантационное генетическое тестирование на анеуплоидии (ПГТ-А). Внедрение ПГТ-А позволило снизить вероятность переноса анеуплоидного эмбриона в полость матки, однако около 50% эуплоидных эмбрионов не имплантируются, в то время как влияние ПГТ-А на исходы программ ВРТ в различных группах пациентов является предметом дискуссий [9–13]. Помимо неоспоримых преимуществ, ПГТ-А обладает и рядом недостатков, среди которых: инвазивность, высокая стоимость и трудоемкость, а также наличие ложных результатов, которые объясняются невозможностью точного определения процента мозаичных клеток в эмбрионе на основании анализа лишь нескольких клеток трофэктодермы [14].
Одним из наиболее перспективных направлений репродукции является изучение роли посттранскрипционных регуляторов экспрессии кодирующих белок генов в эмбриогенезе и возможности использования их в качестве биомаркеров качества эмбрионов. Среди них основное место занимают малые некодирующие РНК [15–17], среди которых самым многочисленным классом у человека являются piwiРНК (8438265 видов молекул согласно базе данных piRBase v.2.0) в сравнении с микроРНК (2600 разновидностей молекул согласно miRBase v.22). Отличительной особенностью piwiРНК является регуляция стабильности генома клетки путем подавления активности мобильных генетических элементов – транспозонов [18, 19]. Существующие механизмы контроля степени экспрессии и мобилизации транспозонов тонко регулируются, особенно в клетках зародышевой линии, так как полное подавление экспрессии транспозонов препятствует нормальному развитию гамет и эмбрионов [20].
На сегодняшний день выявлены особенности экспрессии микроРНК в культуральных средах эмбрионов на разных стадиях эмбриогенеза в зависимости от плоидности их клеток, способности к имплантации и исходов программ ВРТ [21, 22]. Однако до настоящего времени роль piwiРНК в качестве маркера плоидности клеток эмбриона с целью выбора наиболее качественной бластоцисты для переноса в полость матки не была изучена, за исключением наших предыдущих исследований прогностической значимости данного класса молекул в оценке имплантационного потенциала эмбриона на стадии морулы и бластоцисты без учета кариотипа их клеток [23, 24].
В связи с этим целью настоящей работы был анализ содержания piwiРНК в среде культивирования бластоцисты с охарактеризованным кариотипом по данным ПГТ-А для идентификации молекул, ассоциированных с плоидностью клеток эмбриона, и оптимизации селективного переноса эмбриона в рамках программ ВРТ.
Материалы и методы
В исследовании участвовали 343 супружеских пары, обратившиеся для проведения программы ЭКО/ИКСИ+ПГТ-А (экстракорпоральное оплодотворение с интрацитоплазматической инъекцией сперматозоида и последующее проведение ПГТ-А) в НМИЦ АГП им. В.И. Кулакова в период с 2018 по 2021 гг. Было проанализировано 400 циклов ЭКО/ИКСИ+ПГТ-А и 309 криопротоколов. Из 2046 эмбрионов 5-х суток культивирования 1115 эмбрионам было проведено ПГТ-А.
Критериями включения в исследование послужили: отсутствие наступления беременности в течение 1 года и более, нормальный кариотип супругов, наличие таких показаний для проведения ПГТ-А, как старший репродуктивный возраст женщины (35 лет и более), привычный выкидыш (2 и более самопроизвольных прерываний беременности на сроке до 22 недель в анамнезе), повторные неудачные попытки ВРТ (3 и более неудачных попыток селективного переноса «свежих» или размороженных эмбрионов) или тяжелая патозооспермия у партнера (концентрация сперматозоидов – менее 5 млн/мл, количество прогрессивно подвижных форм сперматозоидов (а+b) – 19% и менее, количество морфологически нормальных форм сперматозоидов –1% и менее по результатам спермограммы).
Критериями невключения в исследование послужили все состояния, являющиеся противопоказаниями к проведению ВРТ и беременности в соответствии с приказом МЗ РФ от 31 июля 2020 г. № 803н.
Всем супружеским парам, обратившимся для проведения программы ЭКО/ИКСИ+ПГТ-А, проведено полное клинико-лабораторное обследование.
Гормональная стимуляция яичников проводилась по стандартным протоколам с антагонистом гонадотропин-рилизинг-гормона (антГнРГ) или с агонистом гонадотропин-рилизинг-гормона (аГнРГ). Трансвагинальная пункция яичников проводилась в асептических условиях под внутривенным наркозом через 35–36 ч после введения триггера овуляции. Оплодотворение проводилось методом ЭКО или ИКСИ в зависимости от показателей спермограммы и анамнеза супружеской пары.
Все эмбрионы культивировались в индивидуальных каплях в мультигазовых инкубаторах. Оценка качества полученных эмбрионов проводилась на 5-е сутки культивирования на основании морфологических критериев в соответствии с классификацией Гарднера (ESHRE 2011 «модифицированная» классификация D. Gardner). Образцы культуральных сред эмбрионов, пригодных для ПГТ-А, переносили в пробирки типа Eppendorf и криоконсервировали. У эмбрионов хорошего и отличного качества проводилась биопсия клеток трофэктодермы с последующей криоконсервацией эмбриона. Полученные клетки трофэктодермы переносили в пробирки типа Eppendorf, содержащие лизирующий буфер. ПГТ-А проводили методом высокопроизводительного секвенирования ДНК нового поколения (NGS, next-generation sequencing). Эуплоидный по результатам ПГТ-А эмбрион переносили в полость матки в рамках криопротокола на фоне циклической гормональной терапии или в естественном менструальном цикле при достижении толщины эндометрия более 7 мм. На 10-й день после переноса эмбриона в полость матки определяли уровень β-хорионического гонадотропина человека (ХГч) в сыворотке крови. Для диагностики клинической беременности при положительном результате анализа крови на β-ХГч через 21 день после переноса выполняли трансвагинальное ультразвуковое исследование. Дальнейшее наблюдение и ведение беременности осуществлялись в индивидуальном порядке лечащим врачом.
По результатам ПГТ-А и исходов программ ВРТ были отобраны 73 супружеские пары, у которых были проанализированы 93 эмбриона, входящие в состав следующих групп:
I группа (n=53) включала эуплоидные эмбрионы и была разделена на две подгруппы: Ia группу (n=27) составили эмбрионы, перенос которых привел к имплантации и впоследствии к родам, Ib группа (n=26) – отсутствие имплантации после переноса в полость матки;
II группа (n=40) – анеуплоидные эмбрионы (числовые или структурные аномалии по одной хромосоме – в 65% случаев, по двум хромосомам – в 30% случаев, по трем хромосомам – в 5% случаев; среди хромосомных нарушений преобладали числовые аномалии (53%); структурные изменения хромосом обнаружены в 33%; значительно реже – сочетанные изменения (14%); анеуплоидия выявлена по 3–7, 10–19, 21, 22, Х хромосомам).
В качестве контроля были использованы культуральные среды (n=3) без культивирования в них эмбрионов в течение 5 суток.
Выделение РНК из образцов культуральных сред
Из собранных 96 образцов культуральных сред были выделены РНК колоночным способом с использованием набора miRNeasy Serum/Plasma Kit (Qiagen).
Глубокое секвенирование малых некодирующих РНК
Шесть из 14 мкл элюата колонки miRNeasy Serum/Plasma Kit (Qiagen), содержащего РНК из культуральной среды, были использованы для синтеза кДНК-библиотек набором NEBNext Multiplex Small RNA Library Prep Set for Illumina (Set11, New England Biolab), амплифицированных в течение 30 циклов в ходе полимеразной цепной реакции (ПЦР), очищенных в 6% полиакриламидном геле и секвенированных на платформе NextSeq 500/550 (Illumina). Секвенированные последовательности в диапазоне от 16 до 50 п.н. были картированы на базы данных человека GRCh38.p15, miRBase v21 и piRNABase с использованием алгоритма Bowtie [25]. Анализ дифференциальной экспрессии piwiРНК проводили с помощью программного пакета DESeq2 [26].
Обратная транскрипция и количественная полимеразная цепная реакция в реальном времени
Пять из 14 мкл элюата колонки miRNeasy Serum/Plasma Kit (Qiagen), содержащего РНК из среды культивирования, были использованы для синтеза кДНК набором miScript II RT Kit (Qiagen) по протоколу фирмы-производителя. Количественная ПЦР в реальном времени была проведена с использованием набора miScript SYBR Green PCR Kit (Qiagen, Hilden, Germany) и смысловых праймеров, специфичных для piwiРНК (табл. 1). Программа ПЦР: (1) 15 мин. при 95°С и (2) 40 циклов: 94°С в течение 15 с, оптимизированная температура отжига (45–61°С) в течение 30 с и 70°С в течение 30 с; (3) нагревание реакционной смеси от 65 до 95°C с шагом 0,1°C для построения кривой плавления продукта ПЦР в термоциклере StepOnePlus (Applied Biosystems). Относительную экспрессию piwiРНК в среде культивирования определяли методом ∆∆Ct, используя hsa_piR_022258 в качестве референсной РНК и среду без культивирования эмбриона в качестве контрольного образца.
Статистический анализ
Для статистической обработки результатов использовали скрипты, написанные на языке R [27] и программу RStudio [28]. Соответствие анализируемых параметров закону нормального распределения оценивали по значениям теста Шапиро–Уилка. Статистический анализ проводили с помощью U-критерия Манна–Уитни при сравнении независимых выборок, когда распределение не соответствовало закону нормального распределения. При распределении признаков, отличающемся от нормального, их описывали в виде медианы (Me) и квартилей Q1 и Q3 в формате Me (Q1; Q3). Величину порогового уровня значимости p принимали равной 0,05. Если значение p было меньше 0,001, то p указывали в формате p<0,001. Для описания категориальных бинарных данных использовали абсолютные значения и процентные доли от общего числа в группе и для сравнительного анализа выборок применяли критерий Фишера.
В качестве инструмента для разработки вероятностных моделей наступления того или иного события был выбран метод логистической регрессии. Для оценки качества моделей применялся метод ROC-анализа (Receiver operating characteristic), который является графическим представлением и показывает зависимость истинно положительных примеров от ложноотрицательных. Для предложенных математико-статистических моделей были найдены точки отсечения (пороговые значения), демонстрирующие наиболее адекватное обособление одной группы от другой.
Модели логистической регрессии (линейная регрессия с логит-преобразованием) разрабатывали с использованием программы RStudio путем поэтапного включения и исключения piwiРНК-предикторов качества эмбриона в соответствии с их вкладом в модель. Прогностическую способность модели оценивали методом ROC-анализа по величине AUC (Area Under Curve), статистической значимости, уровню специфичности и чувствительности.
Результаты
73 супружеские пары, включенные в исследование, были подобраны таким образом, чтобы минимизировать влияние вмешивающихся факторов на результаты программ ВРТ. Оценка клинико-лабораторных данных супружеских пар в исследуемых группах не выявила статистически значимых различий, за исключением более длительного периода бесплодия в группе Ia в сравнении с группой Ib (табл. 2). Супружеские пары с отсутствием имплантации при наличии эуплоидного эмбриона характеризовались более низкой концентрацией сперматозоидов по сравнению с супружескими парами, у которых после переноса бластоцисты беременность закончилась своевременными родами (р=0,06). Группа II супружеских пар, у которых был проанализирован анеуплоидный эмбрион, статистически значимо отличалась от группы I супружеских пар, у которых был проанализирован эуплоидный эмбрион по сниженному количеству зрелых ооцитов (М2) относительно общего количества ооцит-кумулюсных комплексов (ОКК) (р=0,03) и повышенному количеству анеуплоидных бластоцист относительно общего количества бластоцист (p<0,001). Надо отметить, что внутри группы II отмечается закономерность повышения доли анеуплоидных бластоцист при снижении процента зрелых ооцитов, что согласуется с литературными данными о возникновении анеуплоидий преимущественно вследствие нарушений оогенеза [29].
При анализе особенностей менструального цикла, овариального резерва и гинекологического анамнеза в группах статистически значимых различий не выявлено.
С целью идентификации малых некодирующих РНК, ассоциированных с анеуплоидией эмбриона, было проведено глубокое секвенирование РНК, выделенной из 20 образцов среды культивирования бластоцисты: 8 эуплоидных эмбрионов и 12 анеуплоидных эмбрионов по данным ПГТ-А (seq(22)x1; seq(22)x1;seq(22)x3;seq(7p)x3,(22)x3; seq(5)x1, (7)x1,6; seq(7)x1, (14)x1; seq(12)x3, (16)x3, (21)x3; seq(7,22)x1, (16)x3; seq(18)x1; seq(19)x3;seq(14)x3;seq(12)x3,(22)x1). Ввиду важной роли piwiРНК в регуляции стабильности генома клеток человека, предметом настоящего исследования явился именно данный тип малых некодирующих РНК. Всего было идентифицировано 133 вида piwiРНК с числами прочтения не менее 10.
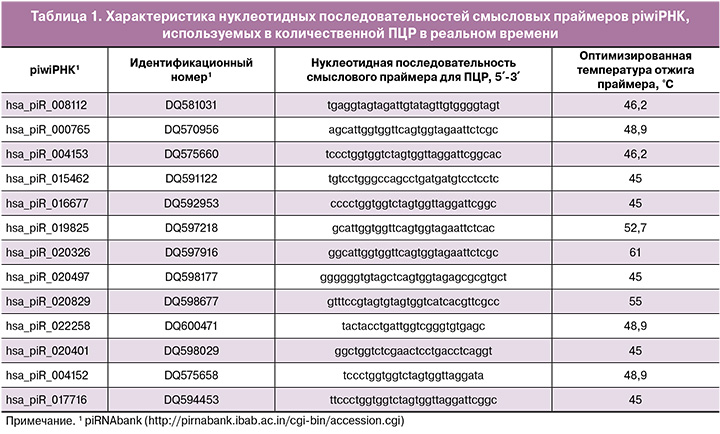
В программе RStudio путем поэтапного включения и исключения каждой идентифицированной молекулы piwiРНК были найдены оптимальные комбинации РНК-маркеров анеуплоидного эмбриона в соответствии с их вкладом в построение моделей логистической регрессии (рис. 1), где в качестве зависимой переменной (переменной отклика) выступала плоидность эмбриона (0 – эуплоидный эмбрион, перенесенный в полость матки с дальнейшим развитием беременности и родов; 1 – анеуплоидный эмбрион, не подлежащий переносу в полость матки). Идентифицировано 12 piwiРНК, которые в различных комбинациях в моделях бинарной логистической регрессии (р<0,001) с высокой чувствительностью (83–100%) и специфичностью (78–100%) диагностировали эуплоидный эмбрион.
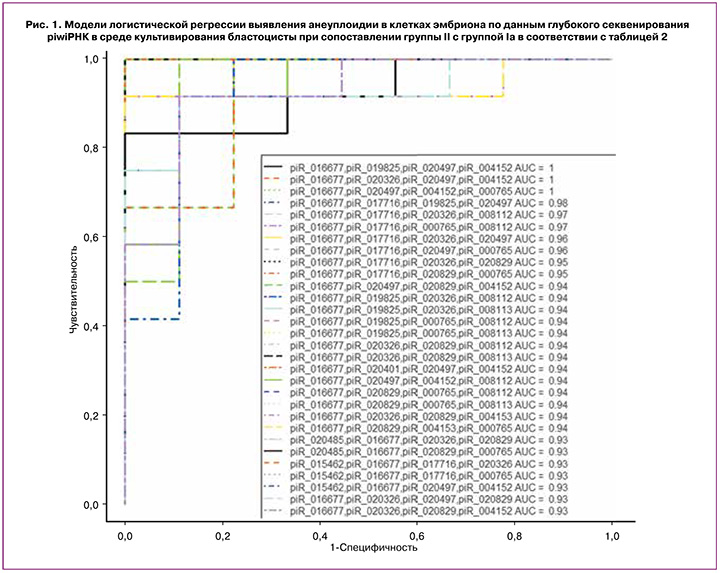
Данные секвенирования были валидированы методом количественной ПЦР в реальном времени на всей выборке из 93 образцов, указанных в таблице 2, и 3 контрольных образцов среды без культивирования в ней эмбриона. По значениям «-ΔΔСt» для каждой из piwiРНК, перечисленных в таблице 1, в программе RStudio была построена бокс-диаграмма содержания piwiРНК в каждой из исследуемых групп (рис. 2).

На бокс-диаграммах, представленных на рисунке 2, по оси ординат отложены значения логарифма по основанию 2-кратности изменения piwiРНК в культуральной среде анализируемых групп бластоцист. Обнаружено статистически значимое повышение уровня piR_004152, piR_015462, piR_020497 в среде культивирования эуплоидных эмбрионов с отрицательным результатом программы ВРТ относительно группы эуплоидных эмбрионов с наличием имплантации (табл. 3, сравнение групп Ia и Ib). В среде культивирования анеуплоидных эмбрионов зафиксировано статистически значимое повышение уровней piR_015462, piR_016677, piR_020497 относительно группы эуплоидных эмбрионов с наличием имплантации (табл. 3, сравнение групп Ia и II). Важно отметить, что группа эуплоидных бластоцист, способных имплантироваться в ткань эндометрия, статистически значимо отличается от любых других бластоцист (эуплоидных эмбрионов без имплантационного потенциала и анеуплоидных бластоцист, не подлежащих переносу в полость матки) по сниженному уровню piR_015462 и piR_020497 в среде их культивирования (табл. 3, сравнение групп Ia и Ib+II). Примечательным фактом является выявленная нами ранее взаимосвязь между уровнем piR_020497 в фолликулярной жидкости и качеством ооцита, способного к оплодотворению и формированию бластоцисты с высоким имплантационным потенциалом [30].

С целью проверки значимости piwiРНК, перечисленных в таблице 3, с точки зрения оценки качества бластоцисты (эуплоидный набор хромосом в бластомерах и высокий имплантационный потенциал) были построены модели логистической регрессии. В программе RStudio путем поэтапного включения и исключения каждой молекулы были найдены оптимальные комбинации piwiРНК, ассоциированные с качеством эмбриона, в соответствии с их вкладом в построение моделей логистической регрессии (рис. 3), где в качестве зависимой переменной (переменной отклика) выступало качество бластоцисты (0 – эуплоидный эмбрион, перенесенный в полость матки с дальнейшим развитием беременности и родов; 1 – анеуплоидный эмбрион, не подлежащий переносу в полость матки).
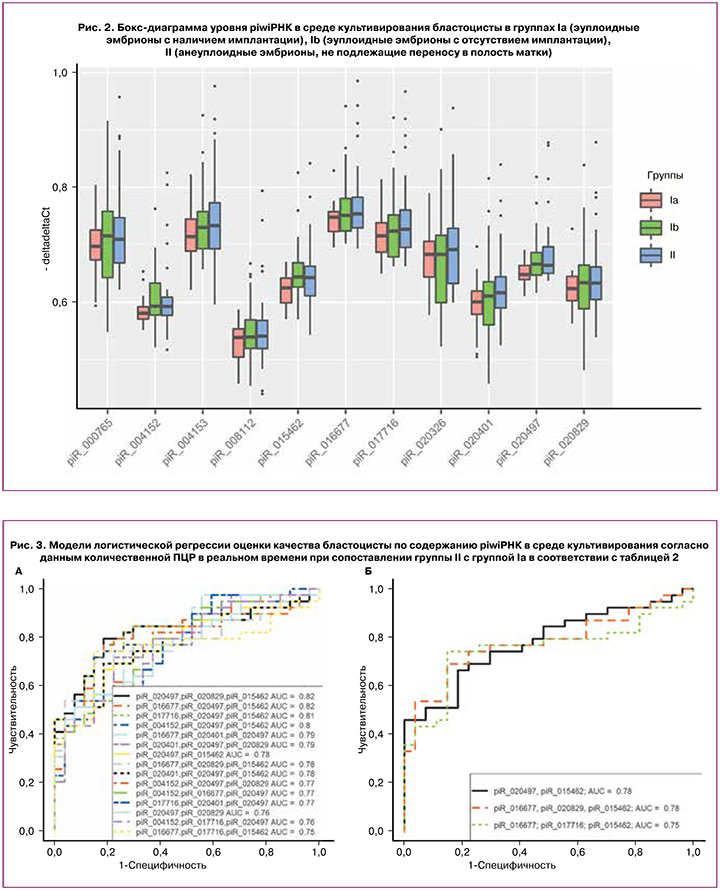
На рисунке 3А представлены все возможные комбинации piwiРНК, участвующие в идентификации эуплоидного эмбриона с высоким имплантационным потенциалом, из которых были отобраны модели, представленные на рисунке 3Б, где все независимые переменные были статистически значимы. Параметры моделей рисунка 3Б указаны в таблице 4. Формулы 1, 2 и 3, описывающие модели рисунка 3Б, представлены ниже.
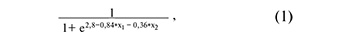
где х1 – значение «-∆∆Сt» для piR_020497, х2 – значение «-∆∆Сt» для piR_015462;
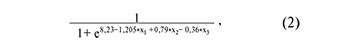
где х1 – значение «-∆∆Сt» для piR_016677, х2 – значение «-∆∆Сt» для piR_020829, х3 – значение «-∆∆Сt» для piR_015462;
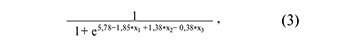
где х1 – значение «-∆∆Сt» для piR_016677, х2 – значение «-∆∆Сt» для piR_017716, х3 – значение «-∆∆Сt» для piR_015462.
Модели рисунка 3Б обладали высокой специфичностью (81–85%), а значит, высокой диагностической ценностью по выявлению эуплоидных бластоцист с высоким имплантационным потенциалом. Более низкие значения чувствительности построенных моделей (67–74%) могут быть обусловлены отсутствием информации об имплантационном потенциале анеуплоидного эмбриона в связи с противопоказанием к переносу в полость матки.
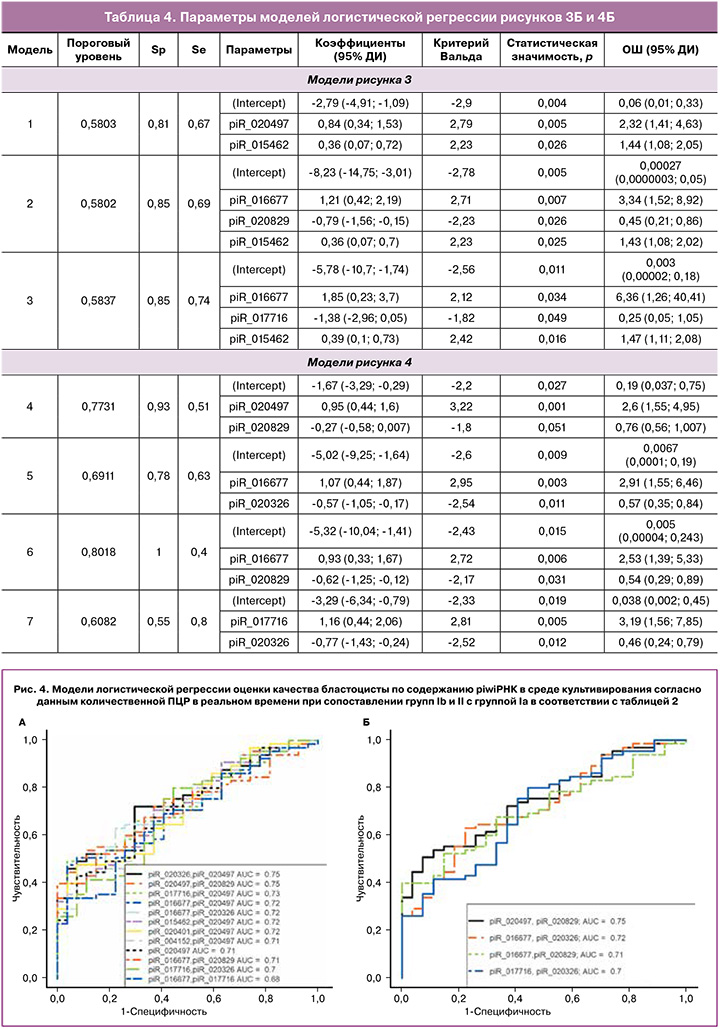
Поскольку не всегда эуплоидный эмбрион обладает высоким имплантационным потенциалом, то для повышения вероятности отбора наиболее качественной бластоцисты для переноса в полость матки с учетом двух факторов – плоидности клеток эмбриона и его имплантационного потенциала – было решено построить модели логистической регрессии при сопоставлении группы Ia c объединенными группами «Ib+II». В программе RStudio путем поэтапного включения и исключения каждой молекулы были найдены оптимальные комбинации piwiРНК в соответствии с их вкладом в построение моделей логистической регрессии (рис. 4), где в качестве зависимой переменной (переменной отклика) выступало качество бластоцисты (0 – эуплоидный эмбрион, перенесенный в полость матки с дальнейшим развитием беременности и родов; 1 – некачественный эмбрион: анеуплоидный эмбрион, не подлежащий переносу в полость матки, и эуплоидный эмбрион с отсутствием имплантации после переноса в полость матки).
На рисунке 4А представлены все возможные комбинации piwiРНК, участвующие в выявлении качественной бластоцисты, из которых были отобраны модели, представленные на рисунке 4Б, где все независимые переменные были статистически значимы. Параметры моделей рисунка 4Б указаны в таблице 4. Формулы 4, 5, 6 и 7, описывающие модели рисунка 4Б, представлены ниже.
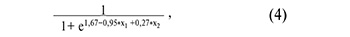
где х1 – значение «-∆∆Сt» для piR_020497, х2 – значение «-∆∆Сt» для piR_020829;
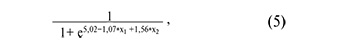
где х1 – значение «-∆∆Сt» для piR_016677, х2 – значение «-∆∆Сt» для piR_020326;
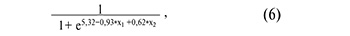
где х1 – значение «-∆∆Сt» для piR_016677, х2 – значение «-∆∆Сt» для piR_020829;
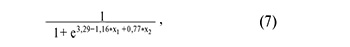
где х1 – значение «-∆∆Сt» для piR_017716, х2 – значение «-∆∆Сt» для piR_020326.
Наибольшей диагностической значимостью с точки зрения выявления качественного эмбриона (эуплоидный с высоким имплантационным потенциалом) обладают модели 4 и 6 рисунка 4Б ввиду наибольшей специфичности (93 и 100% соответственно) при сравнении с моделями 5 и 7 рисунка 4Б. Более низкую чувствительность моделей 4 и 6 (51 и 40% соответственно) в сравнении с их специфичностью можно объяснить отсутствием информации об имплантационном потенциале анеуплоидного эмбриона в связи с противопоказанием к переносу в полость матки и отсутствием информации о рецептивности эндометрия на момент переноса эуплоидного эмбриона в полость матки.
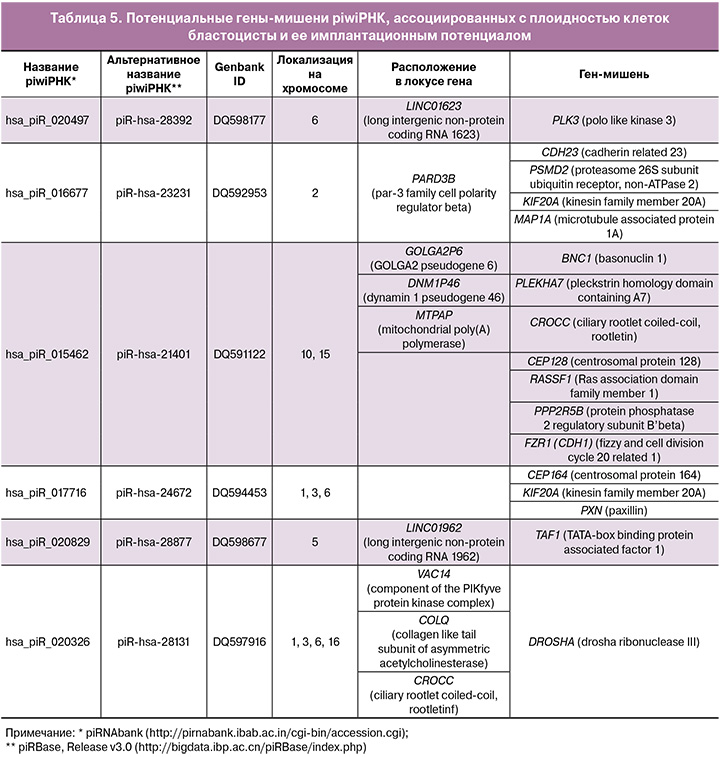
С целью анализа функциональной значимости piwiРНК, входящих в состав моделей рисунка 3Б и рисунка 4Б, были идентифицированы потенциальные гены-мишени piwiРНК с использованием базы данных GRCh38, содержащей последовательности кодирующих белок транскриптов (https://www.ncbi.nlm.nih.gov/genome/guide/human/) и алгоритма miRanda со следующими параметрами: счет выравнивания sc≥170 и энергия связывания
en≤ -20.0 kcal mol-1, как описано в нашем недавнем исследовании [24]. Среди десятков тысяч идентифицированных генов-мишеней для некоторых из них было выявлено их участие в регуляции формирования веретена деления клетки, функционирования кинетохора и цитокинеза, нарушения которых являются причиной анеуплоидии (табл. 5).
Обсуждение
В настоящем исследовании при использовании метода глубокого секвенирования малых некодирующих РНК с последующей валидацией полученных данных методом количественной ПЦР в реальном времени были получены модели логистической регрессии по идентификации эуплоидной бластоцисты с высоким имплантационным потенциалом по уровню piwiРНК в среде культивирования эмбриона на 5-е сутки после оплодотворения, среди которых количественная оценка комбинации piR_020497 и piR_020829 либо piR_016677 и piR_020829 обладала наибольшей специфичностью (93 и 100% соответственно), а значит, диагностической ценностью при отборе наиболее качественной бластоцисты для переноса в полость матки с учетом двух факторов – плоидности клеток эмбриона и его имплантационного потенциала. Очевидно, что для применения разработанных моделей в клинической практике необходима их валидация на многочисленных тестовых выборках с подтверждением плоидности клеток эмбриона методом ПГТ-А. Кроме того, для повышения точности работы разработанных моделей необходима оценка рецептивности эндометрия в день переноса криоконсервированного эмбриона. В случае получения доказательств надежности и эффективности разработанных моделей появится возможность не прибегать к инвазивному методу ПГТ-А с целью проведения скринингового теста оценки качества всех полученных у супружеской пары эмбрионов.
Одной из функций piwiРНК является регуляция стабильности генома клетки путем взаимодействия в ядре с транскриптом ретротранспозона в составе комплекса RISC (РНК-индуцируемый комплекс сайленсинга), контактирующего с деацетилазой и метилтрансферазой гистонов и/или ДНК-метилтрансферазой, которые блокируют дальнейшую транскрипцию ретротранспозона, тем самым препятствуя его активности и встраиванию в различные участки генома [17]. В работе Krawetz S.A. et al. были обнаружены piwiРНК сперматозоидов, которые имели потенциальную аффинность связывания с определенными транспозонами [31]. Среди идентифицированных нами piwiРНК в среде культивирования бластоцисты две молекулы, hsa_piR_020829 и hsa_piR_017716, по данным этого исследования, экспрессировались в сперматозоидах, а для hsa_piR_020829 были идентифицированы места связывания с ретротранспозонами L1 (длинные диспергированные ядерные повторы) и MER (умеренно повторяющиеся диспергированные последовательности).
ДНК-последовательности определенных классов piwiРНК расположены внутри белок-кодирующих генов, и функционально активные молекулы piwiРНК в основном продуцируются из 3’-нетранслируемых областей (3’-НТО) мРНК во время их трансляции [17]. Кроме того, 3’-НТО мРНК, продуцирующие piwiРНК, как правило, содержат последовательности транспозонов, активность которых контролируется данными piwiРНК на посттранскрипционном уровне, что было продемонстрировано во время постмейотических стадий сперматогенеза [32]. Согласованные процессы трансляции мРНК и одновременного образования функциональных piwiРНК, нацеленных на снижение активности транспозонов, являются проявлением тонкой регуляции гаметогенеза, от которой в дальнейшем зависит качество образуемого эмбриона. Некоторые piwiРНК, идентифицированные в настоящем исследовании как маркеры эуплоидной бластоцисты с высоким имплантационным потенциалом, расположены в локусах белок-кодирующих генов, а именно: ДНК-последовательность hsa_piR_016677 расположена в гене PARD3B, hsa_piR_015462 – в генах DNM1P46 и MTPAP, hsa_piR_020326 – в генах VAC14, COLQ и CROCC.
Помимо супрессорной активности piwiРНК в отношении транспозонов, также известно об их регуляторном воздействии на сигнальные пути в клетке посредством различных молекулярных механизмов [17]. Например, пахитенные piwiРНК направляют белок MIWI к мРНК-мишени либо для взаимодействия с деаденилазой CAF1, осуществляющей деаденилирование и последующую деградацию мРНК [33], либо для прямого расщепления мРНК [34, 35]. Данные механизмы по удалению огромного числа мРНК-мишеней, например, работают на стадии поздних сперматид перед началом их морфологической трансформации в сперматозоиды. Парадоксально, но piwiРНК могут как дестабилизировать, так и стабилизировать мРНК-мишень, активируя ее трансляцию. Например, недавнее исследование показало, что пахитенные piwiРНК в комплексе с MIWI, субъединицей F фактора инициации трансляции 3 (eIF3F) и РНК-связывающим белком HuR активируют трансляцию ряда спермиогенных мРНК в круглых сперматидах, тем самым детерминируя определенную стадию гаметогенеза [36]. При использовании алгоритма miRanda нами были идентифицированы потенциальные гены-мишени piwiРНК, являющихся маркерами эуплоидной бластоцисты с высоким имплантационном потенциалом. Важно отметить, что белковые продукты данных генов-мишеней участвуют в формировании веретена деления, образовании и функционировании кинетохора, цитокинезе, нарушения которых приводят к возникновению анеуплоидии клетки.
Так, например, Polo-подобная киназа 3 (PLK3) – ген-мишень hsa_piR_020497 – локализуется в области ядра, центросомы и борозды деления клетки и участвует в регуляции клеточного цикла путем активации CDC25А и CDC25C, индуцируя G1/S- и G2/M-переход соответственно, и в цитокинезе [37]. Филогенетический анализ показал высокую консервативность киназного и белок-связывающего домена у PLK1-3, поэтому структурное сходство этих киназ обусловливает одинаковую функциональную активность. Доказано участие PLK1 в разъединении центросом путем поддержания активности протеинкиназы NEK2A, фосфорилирующей линкер двух центросом – рутлетин (CROCC, ген-мишень hsa_piR_015462). В случае мутаций в гене CROCC возникают выраженная хромосомная нестабильность и нарушения сегрегации хромосом [38]. Участие PLK1–3 в цитокинезе происходит за счет взаимодействия с различными субстратами, в том числе с белком семейства кинезинов (KIF20A, ген-мишень hsa_piR_016677 и hsa_piR_017716), которые и обеспечивают перемещение PLK1-3 из центросом и кинетохор в среднюю зону веретена деления за счет катализа быстрого пространственного ремоделирования микротрубочек, необходимого для внутриклеточного транспорта белков и органелл [39]. Кроме того, ряд субъединиц основной фосфатазы, ассоциированных с микротрубочками белков, – РР2А (PPP2R1A, PPP2R2D, PPP2R3B, PPP2R5B и PPP2R5D) также взаимодействуют с PLK1 и участвуют в поддержании геномной стабильности [40]. Продемонстрировано, что ингибирование PPP2R5B (ген-мишень hsa_piR_015462) воздействует на сцепление сестринских хроматид в клетках с повышенным уровнем экспрессии PLK1. SET/TAF1 (ген-мишень hsa_piR_020829) является ингибитором РР2А, локализуется в центромерах и поддерживает активность киназы Aurora B, которая фосфорилирует различные компоненты кинетохора для коррекции неправильного соединения кинетохора с микротрубочками. Своевременное высвобождение SET из кинетохорного белкового комплекса во время метафазы обеспечивает стабилизацию соединения кинетохора с микротрубочками под действием фосфатазной активности РР2А [41].
Центросома является сайтом концентрации протеасомы и ассоциированных регуляторных белков [42]. В клетках эукариот деградация 80–90% белков осуществляется протеасомой 26S [43], состоящей из двух комплексов – корового 20S и регуляторного 19S, в котором PSMD2 (ген-мишень hsa_piR_016677) кодирует одну из субъединиц [44]. Важно отметить, что Polo-подобные киназы подвергаются убиквитин-зависимому протеолизу в протеасоме [37], что в случае регуляции концентрации PSMD2 и PLK3 под действием hsa_piR_016677 и piR_020497 соответственно подчеркивает сложную систему контроля формирования структур веретена деления для правильной сегрегации хромосом. Другим важным регулятором клеточного цикла с использованием убиквитин-опосредованного протеолиза является индуцирующий анафазу комплекс или циклосома APC/C, активирующаяся Cdc20 или Cdh1 (ген-мишень hsa_piR_015462). Во время оогенеза APC/CCdh1 путем деградации циклина В1 обеспечивает поддержание пролонгированного ареста примордиальных фолликулов на стадии профазы I мейоза и блокирование их вхождения в метафазу I [45]. На стадии анафазы I APC/CCdh1 обеспечивает фосфорилирование связанного с центромерами хромосом Sgol2 (shugoshin-подобного белка 2) под действием киназы Aurora B/C, что приводит к диссоциации Sgol2 и расхождению хромосом [46]. В случае делеции генов Apc и Cdh1 в ооците Sgol2 остается связанным с хромосомами в анафазе I, что не приводит к их расхождению и обусловливает возникновение анеуплоидий после первого мейотического деления.
Выявленные нами piwiРНК, ассоциированные с анеуплоидией клеток бластоцисты, потенциально регулируют уровень центриолярных белков, играющих важную роль в оогенезе и сперматогенезе. СEP128 (ген-мишень hsa_piR_015462), CEP164 (ген-мишень hsa_piR_017716) и базонуклин 1 (BNC1, ген-мишень hsa_piR_015462) являются ключевыми белками придатка материнской центриоли мужских половых клеток, участвуя в организации центриолярных микротрубочек и формировании аксонемы, а в случае дефектов кодирующих их генов происходят нарушения сперматогенеза и фертилизации вследствие снижения концентрации и морфологии сперматозоидов [47–50]. Роль BNC1 в репродукции была продемонстрирована не только с позиций регуляции сперматогенеза, но и оогенеза: в случае ингибирования экспрессии гена BNC1 в ооците происходила фертилизация, но развитие эмбриона останавливалось на двухклеточной стадии [51].
На доимплантационном этапе эмбриогенеза, начиная с 8-клеточной стадии, бластомеры подвергаются поляризации и компактизации путем реорганизации микротрубочек, актиновых и промежуточных филаментов, четкая и своевременная регуляция которой обусловливает формирование эмбриобласта и трофобласта бластоцисты, способной к имплантации [52]. При миграции клеток растущие концы микротрубочек заякориваются в участках фокальной адгезии, что приводит к дестабилизации адгезивного контакта и откреплению клетки от субстрата. Основную роль в данном процессе играет белок фокальной адгезии паксиллин (PXN, ген-мишень hsa_piR_017716) путем взаимодействия с α-тубулином [53]. Продемонстрировано значительное повышение экспрессии PXN в области образования фокальных контактов после компактизации и бластуляции эмбриона для формирования адгезивных свойств бластоцисты и обеспечения имплантации [54]. Белок адгезии CDH23 (ген-мишень hsa_piR_016677) является регулятором сети микротрубочек, поддерживая полимеризацию “-” конца микротрубочек путем взаимодействия с белком CAMSAP3 [55], при этом обеспечивая стабильность адгезионного межклеточного контакта при взаимодействии с белком PLEKHA7 (ген-мишень hsa_piR_015462) [56]. Примечательно, что PLEKHA7 участвует не только в стабилизации основных трансмембранных компонентов адгезионного межклеточного контакта, но и взаимодействует с ферментным комплексом процессинга микроРНК (DROSHA и DGCR8), обеспечивая регуляцию miR-24, miR-30a, miR30-b и let7-g, которые подавляют экспрессию индуцирующих клеточный рост белков SNAIL, MYC и циклина D1 непосредственно в местах межклеточного контакта [57]. Надо отметить, что потенциальным геном-мишенью молекулы hsa_piR_020326, идентифицированной нами как один из маркеров анеуплоидии клеток эмбриона, является фермент DROSHA, что подчеркивает сложную взаимосвязь разных видов малых некодирующих РНК (piwiРНК и микроРНК) и белок-кодирующих РНК, при дисбалансе которых возникает та или иная патология.
Важную роль в сперматогенезе играет белок MAP1A (ген-мишень hsa_piR_016677), ассоциированный с микротрубочками клеток Сертоли эпителиосперматогенного слоя семенных канальцев [58] и стабилизирующий микротрубочковую структуру направленного транспорта дифференцирующихся гамет от наружной базальной части стенки канальцев, содержащей сперматогонии, до внутренней адлюминальной части, содержащей сперматоциты, сперматиды и сперматозоиды. Любое нарушение эпителиального цикла сперматогенеза, в том числе вследствие изменения уровня экспрессии MAP1A, приведет к снижению мужской фертильности. Нельзя не отметить ключевую роль белка RASSF1A (ген-мишень hsa_piR_015462) в стабилизации микротрубочек посредством ингибиторного воздействия на деацетилазу HDAC6, способствуя ацетилированию микротрубочек – стабильной, долгоживущей структуры, необходимой для направленного транспорта везикул, формирования центросомы и аппарата Гольджи, стабилизации участков фокальной адгезии и межклеточных контактов [59].
Заключение
Таким образом, анализ литературных данных о функции потенциальных генов-мишеней определенного набора молекул piwiРНК, идентифицированных в настоящем исследовании как маркеры плоидности эмбриона и его имплантационного потенциала, позволяют заключить, что качество эмбриона зависит от четко координированного в пространстве и времени гаметогенеза, где постоянство числа хромосом при каждом клеточном делении зависит от правильного формирования веретена деления, обеспечивающего равноценную для обеих дочерних клеток сегрегацию хромосом, где одними из ключевых регуляторных молекул выступают piwiРНК. В связи с этим, в настоящем исследовании предложен неинвазивный способ идентификации эуплоидного эмбриона с высоким имплантационным потенциалом на основе количественной оценки piwiРНК в среде его культивирования, позволяющий с высокой специфичностью (93–100%) осуществлять выбор эмбриона для переноса в полость матки. Разработанный в настоящей работе метод необходимо апробировать на нескольких тестовых выборках для внедрения в клиническую практику. Для экспериментального подтверждения взаимосвязи дисбаланса уровня экспрессии piwiРНК и их генов-мишеней целесообразно дальнейшее проведение транскриптомного исследования белок-кодирующих генов генома эмбриона в группах анеуплоидных бластоцист и эуплоидных бластоцист плохого качества, не подлежащих переносу в полость матки.



