Наиболее наглядной клинической моделью нарушений репродуктивной системы является бесплодие неясного генеза (БНГ), при котором репродуктивная система анатомически и функционально полноценна, однако беременность не наступает [1–3].
По данным литературы, проводимые в мире исследования, посвященные изучению БНГ, немногочисленны и обладают невысоким уровнем доказательности. Международным консенсусом были определены 10 приоритетных направлений будущих исследований в отношении бесплодия, в их числе – БНГ [4].
Ранее мы получили данные о том, что при БНГ в программах экстракорпорального оплодотворения (ЭКО) количество зрелых ооцитов достаточное, частота оплодотворения нормальная, однако частота бластуляции низкая, в основном за счет остановок развития эмбрионов до 3-х суток культивирования, что является признаком нарушения раннего эмбриогенеза и, следовательно, показанием для использования ЭКО с целью достижения беременности [5].
Спектр исследований репродуктивной системы человека расширяется и в настоящее время включает высокотехнологичные методы, направленные на получение генетической, эпигенетической и другой информации при различных нарушениях репродуктивной системы [6].
Полноэкзомное секвенирование – один из методов высокопроизводительного секвенирования (next generation sequencing, NGS), с помощью которого одномоментно расшифровывается структура экзонов – последовательностей ДНК, кодирующих белки человека [7].
При проведении полноэкзомного секвенирования могут быть выявлены варианты известных генов, а также обнаруживаться новые генетические особенности и закономерности, возможно, связанные с исследуемым заболеванием. Также полноэкзомное секвенирование может служить исследовательским инструментом поиска при отсутствии гипотезы, то есть предварительного представления о том, какие гены, сигнальные каскады или метаболические пути изменены в организме пациента, как при БНГ. В этом случае в исследование включаются не семейные, а спорадические случаи заболевания с похожей клинической картиной [8, 9].
Проведение полноэкзомного секвенирования у супружеских пар с БНГ позволит проверить наличие вариантов, роль которых обсуждается в научных исследованиях и обнаружить новые особенности и закономерности в генотипе у пациентов с БНГ [10]. Так, низкое качество ооцитов при бесплодии может быть обусловлено вариантами генов PATL2, TUBB8, WEE2 и PAD16: изменение экспрессии генов [OMIM gene ID: 614661, OMIM phenotype: OOMD4 (oocyte maturation defect, нарушение созревания ооцитов), OMIM phenotype ID: 617743, наследование: аутосомно-рецессивное] PATL2 [15q21.1] и [OMIM gene ID: 616768.0001, OMIM phenotype: OOMD2, OMIM phenotype ID: 616780, наследование: аутосомно-доминантное] TUBB8 [10p15.3] приводит к нарушению созревания ооцитов в MII; [OMIM gene ID: 614084.0001-614084.0003, OMIM phenotype: OOMD5, OMIM phenotype ID: 617996, наследование: аутосомно-рецессивное] WEE2 [7q34] – снижению способности ооцитов к полноценному оплодотворению и формированию эмбрионов; [OMIM gene ID: 610363.0001-610363.0005, OMIM phenotype: PREMBL2 (preimplantation embryonic lethality, преимплантационная гибель эмбрионов), OMIM phenotype ID: 617234, наследование: аутосомно-рецессивное] PAD16 [1p36.13] – остановке развития эмбрионов на ранних стадиях. С дефектами созревания ооцитов и преимплантационной эмбриональной летальностью также могут быть связаны гены ZP1, ZP3, ZP2, TLE6 [11, 12].
При проведении полноэкзомного секвенирования помимо основной информации в качестве случайных находок могут обнаруживаться другие значимые с медицинской точки зрения варианты, в связи с чем возникает этический вопрос о необходимости и объеме предоставляемой пациенту информации. Специалистами Американского колледжа медицинской генетики и геномики (American College of Medical Genetics and Genomics, ACMG) опубликован список генов, об обнаружении вариантов которых рекомендуется сообщать пациентам с целью ранней диагностики заболевания, однако единого решения этической проблемы полноэкзомного секвенирования в настоящее время не разработано [13, 14].
Сегодня назначение генетических исследований пациентам с БНГ не регламентировано клиническими рекомендациями и проводится эмпирически при незакономерно малом количестве зрелых ооцитов, низкой частоте оплодотворения, остановке развития эмбрионов на ранних стадиях в программах ЭКО.
Цель исследования: провести полноэкзомное секвенирование супружеских пар с БНГ.
Материалы и методы
БНГ диагностируется у пар, стандартное обследование которых не приводит к установлению причины бесплодия: менструальный цикл регулярный, овуляторный, маточные трубы проходимы, эндометрий без патологических изменений, показатели спермы в норме, при проведении лапароскопии и гистероскопии патология не обнаруживается.
В отделении клинической генетики Института репродуктивной генетики ФГБУ «НМИЦ АГП им. В.И. Кулакова» Минздрава России (далее – Центр) было проведено пилотное исследование – полноэкзомное секвенирование ДНК из образцов периферической крови, полученных от 6 супружеских пар (женщин и мужчин), целенаправленно и методично отобранных из проспективной группы исследования пациентов с БНГ (n=45) для того, чтобы исключить предполагаемые генетические причины бесплодия у супружеских пар с БНГ [3].
Отбор пациентов для полноэкзомного секвенирования проводился согласно критериям:
- диагноз бесплодия установлен до 35 лет;
- длительность бесплодия 5 лет и более;
- неотягощенный соматический и гинекологический анамнез;
- нормальный уровень гормонов в крови;
- нормозооспермия у партнера;
- нормальный женский – (46ХХ) и мужской – (46ХY) кариотипы;
- отсутствие признаков патологии при ультразвуковом исследовании органов и систем;
- безуспешные попытки внутриматочной инсеминации;
- наличие 3 и более безуспешных попыток ЭКО с регистрацией нарушения развития эмбрионов или отсутствием эмбрионов.
Оценка клинической значимости (патогенности) выявленных вариантов проводилась на основе рекомендаций ACMG и рекомендаций Российского общества медицинских генетиков по интерпретации данных, полученных методами высокопроизводительного секвенирования [15, 16].
У включенных в исследование 6 супружеских пар с БНГ проводился забор 25 мл периферической крови из локтевой вены. Все пациенты подписали информированное добровольное согласие об участии в исследовании. Секвенирование ДНК пациентов проводилось на платформе NovaSeq 6000 (WES, секвенирование полного экзома). Для обогащения целевыми фрагментами использовался набор для обогащения Illumina TruSeq Exome.
Обработка данных секвенирования проводилась с использованием алгоритма, включающего выравнивание прочтений на референсную последовательность генома hg38, коллинг и фильтрацию вариантов по качеству. Для всех вариантов, прошедших фильтрацию по качеству, проводилась аннотация с использованием Ensembl Variant Effect Predictor (VEP) с применением ряда алгоритмов предсказания значимости вариантов (SIFT, PolyPhen-2, SpliceAI) [17].
Для оценки популяционных частот выявленных вариантов использовалась база данных Genome Aggregation Database (gnomAD) [18]. Для оценки клинической релевантности выявленных вариантов использовались базы данных OMIM, ClinVar, LOVD, RUSeq и другие специализированные базы данных [19–23].
Рассматривались варианты, с которыми могло быть связано бесплодие в паре, и случайные находки, согласно списку генов для сообщения вторичных находок ACMG, за исключением не описанных вариантов в гене TTN, приводящих к потере функции белка [13, 14, 24].
Следует отметить, что значимость обнаруженных вариантов может меняться с течением времени при накоплении научных и клинических данных.
Ограничения: метод не позволяет выявлять инсерции и делеции длиной более 10 пар нуклеотидов, варианты в интронных областях (за исключением канонических сайтов сплайсинга), вариации длины повторов (в том числе экспансии триплетов). Метод не предназначен для определения цис- или транс-положения пар гетерозиготных вариантов, а также для оценки уровня метилирования, выявления хромосомных перестроек, полиплоидии, вариантов в состоянии мозаицизма [15].
Результаты
По результатам полноэкзомного секвенирования у исследованных супружеских пар не были обнаружены патогенные варианты, имеющие отношение к фенотипу пациентов; вероятно патогенные варианты, имеющие отношение к фенотипу пациентов; случайные (вторичные) находки (согласно списку генов ACMG). В двух супружеских парах были выявлены варианты неясной клинической значимости, описанные ниже.
Супружеская пара № 1: Б., 27 лет (ж), С., 36 лет (м), Москва, национальность: русские. В течение 6 лет половой жизни без предохранения с момента вступления в брак беременность не наступала. Обследовались с 2017 по 2021 гг., причина бесплодия не была установлена. Проходили лечение с 2018 по 2020 гг.: две внутриматочные инсеминации, две программы ЭКО: в естественном цикле и в цикле, стимулированном по «длинному» протоколу с агонистами гонадотропин рилизинг-гормона; беременность не наступила. В 2020 г. было три попытки ЭКО с партнером и одна попытка с использованием донорской спермы – во всех программах эмбрионы останавливались в развитии на 3–4 сутки (табл. 1). Обратились в Центр, 03.03.2021 установлен диагноз: «Бесплодие неясного генеза. Повторные неудачи вспомогательных репродуктивных технологий. Арест раннего эмбриогенеза» и рекомендовано проведение программы ЭКО: получено 12 ооцитов, из них зрелых – 7, оплодотворилось 2 ооцита – 2 зиготы, образовалось 2 бластоцисты, наступила беременность, благополучно завершившаяся родами.
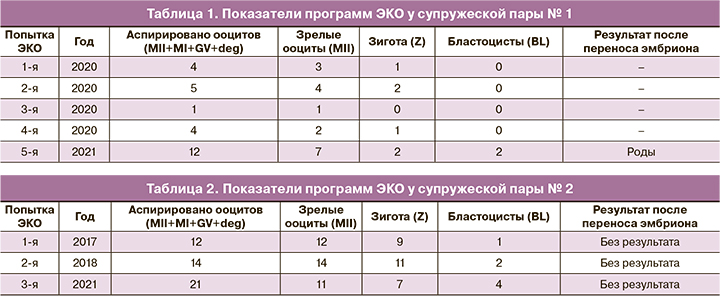
У женщины из данной супружеской пары выявлен вариант неясной клинической значимости: ранее не описанный гетерозиготный вариант нуклеотидной последовательности в гене рецептора ФСГ (FSHR) (2-48963163-A-G), приводящий к замене аминокислоты (p. Tyr553Cys, NM_000145.4). Программы-предсказатели dbNSFP, PolyPhen, Sift классифицируют вариант как патогенный. Варианты в гене FSHR типа миссенс описаны, в том числе, у пациентов с аутосомно-рецессивной дисгенезией яичников (OMIM: 233300, Ovarian dysgenesis 1) и аутосомно-доминантным синдромом гиперстимуляции яичников (OMIM: 608115, Ovarian hyperstimulation syndrome). Вариант не зарегистрирован в контрольной выборке gnomAD и RUSeq.
Супружеская пара № 2: К., 34 года (ж), К., 33 года (м), Москва, национальность: русские. В течение 10 лет половой жизни без предохранения в браке беременность не наступала. В 2015 г. был установлен диагноз: «Бесплодие неясного генеза», была проведена овариальная стимуляция с внутриматочной инсеминацией, беременность не наступила. С 2017 по 2021 гг. было три попытки ЭКО с получением достаточного количества ооцитов (12, 14, 21 ооцитов соответственно), но низким выходом бластоцист (1, 2, 2 бластоцисты соответственно), переносом одной бластоцисты в каждой программе, криопереносом оставшихся бластоцист, пригодных для переноса по данным преимплантационного генетического тестирования на анеуплоидии (ПГТ-А), беременность не наступила (табл. 2).
Супружеская пара № 3: П., 39 лет (ж), П., 40 лет (м), Калужская область, национальность: русские. В течение 5 лет половой жизни без предохранения в браке беременность не наступала. Обследовались с 2012 по 2019 гг., причина бесплодия не была установлена. Было проведено две внутриматочные инсеминации: со стимуляцией овуляции и в естественном цикле, беременность не наступила. В 2019 г. был установлен диагноз: «Бесплодие неясного генеза» и рекомендовано проведение программы ЭКО. С 2019 по 2021 гг. было четыре попытки ЭКО, в трех из которых отмечался низкий выход бластоцист, в результате последней попытки на сроке 5–6 недель была диагностирована левосторонняя трубная беременность – произведена лапароскопия с тубэктомией слева (табл. 3).
Супружеская пара № 4: Б., 38 лет (ж), Д., 40 лет (м), Республика Северная Осетия–Алания, национальность: осетины. В течение 5 лет половой жизни без предохранения в браке беременность наступила один раз в 2017 г., прервалась на сроке 4 недели, без осложнений. В 2020 г. было две попытки ЭКО, беременность не наступила. В 2021 г. проведена третья попытка ЭКО: при достаточном количестве ооцитов (получено 17 ооцитов, из них зрелых – 15) и зигот (10 зигот) бластоцист не образовалось (0 бластоцист) (табл. 4).
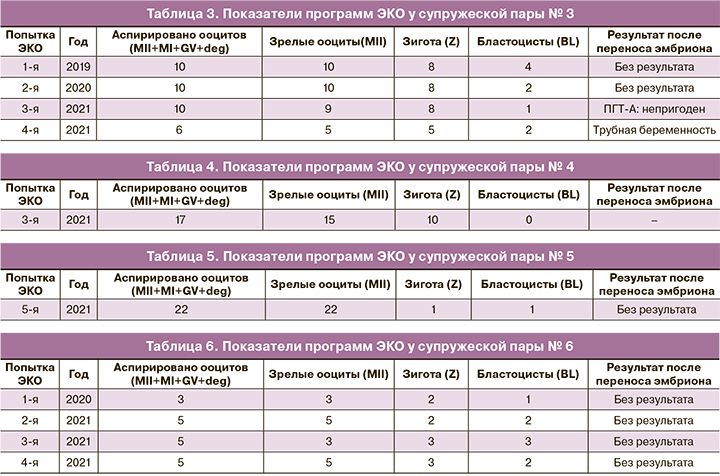
Супружеская пара № 5: Л., 34 года (ж), Л., 36 лет (м), Московская область, национальность: русские. В течение 13 лет половой жизни без предохранения в браке беременность не наступала. Обследовались с 2012 г., причина бесплодия не была установлена. Была проведена овариальная стимуляция с внутриматочной инсеминацией, беременность не наступила. С 2014 по 2021 гг. было четыре попытки ЭКО в разных клиниках с получением достаточного количества ооцитов (17, 24, 20, 20 ооцитов соответственно), удовлетворительным количеством бластоцист (7, 6, 14, 4 бластоцист соответственно), переносом двух бластоцист в трех программах (в четвертой программе перенос не производился в связи с низким качеством эмбрионов), однако беременность не наступила. ПГТ-А не проводилось, потому что пригодных для биопсии эмбрионов не было. В 2021 г. обратились в Центр, было рекомендовано проведение программы ЭКО: получено 22 ооцита, из них зрелых – 22, оплодотворился 1 ооцит – 1 зигота, образовалась 1 бластоциста, беременность не наступила (табл. 5). Ретроспективно установлен диагноз: «Бесплодие неясного генеза. Повторные неудачи ВРТ. Ооцитарный фактор?».
У мужчины выявлен вариант неясной клинической значимости: ранее описанный гетерозиготный вариант нуклеотидной последовательности в гене GNRHR (4-67740682-C-T), приводящий к замене аминокислоты в 262 позиции белка (p.Arg262Gln, NM_000406, rs104893837). Вариант описан в компаунд-гетерозиготной форме с другими вариантами и в гомозиготной форме у пациентов с гипогонадотропным гипогонадизмом (OMIM: 146110, Hypogonadotropic hypogonadism 7 without anosmia; OMIM: 138850#0002). Вариант зарегистрирован в контрольной выборке gnomAD в количестве 505 мутантных аллелей на 282244 хромосомы, в том числе 1 гомозигота, и RUSeq с количеством наблюдений 4/3376, частотой варианта 0.001185. По совокупности сведений, учитывая невыявление второго патогенного варианта в гене, его следует расценивать как вариант с неопределенной клинической значимостью.
Супружеская пара № 6: А., 35 лет (ж), А., 37 лет (м), Москва, национальность: русские. В течение 6 лет половой жизни без предохранения в браке беременность не наступала. Обследовались с 2019 г., причина бесплодия не была установлена. С 2020 по 2021 гг. было три попытки ЭКО, в которых получали мало ооцитов (3, 5, 5 ооцитов соответственно) и сопоставимое количество бластоцист (2, 2, 3 бластоцисты соответственно), проводились перенос 1 бластоцисты, криоперенос 1 бластоцисты, два раза - перенос 2 бластоцист высокого качества, соответственно, беременность не наступила. ПГТ-А не проводилось в связи с отсутствием пригодных для биопсии эмбрионов. В 2021 г. установлен диагноз: «Бесплодие неясного генеза», и рекомендовано проведение четвертой программы ЭКО: получено 5 ооцитов, из них зрелых – 5, оплодотворилось 3 ооцита – 3 зиготы, образовалось 2 бластоцисты высокого качества, обе перенесены, беременность не наступила (таблица 6).
Таким образом, при полноэкзомном секвенировании у пациентов с БНГ не было обнаружено известных вариантов, с которыми может быть связано бесплодие. Необходим дальнейший научный поиск молекулярных механизмов нарушения ранних этапов эмбриогенеза у пациентов с БНГ, включающий в первую очередь исследование качества гамет.
Обсуждение
БНГ диагностируется при отсутствии у пары явных дефектов репродуктивной системы. Ранее были уточнены признаки БНГ: нормальная частота оплодотворения, сниженная частота бластуляции за счет большей частоты остановок развития эмбрионов до 3-х суток культивирования и в то же время более высокое качество получаемых бластоцист. То есть зачатие у этих супружеских пар происходит с нормальной частотой, эмбрионы чаще останавливаются в развитии до 3-х суток, бластоцисты образуются реже, но часто они хорошего качества; поэтому беременность может наступать, но время до ее достижения увеличивается; при этом возраст и увеличивающаяся с возрастом коморбидность пациентов с БНГ приводят к дополнительному ограничению реализации их репродуктивной функции. ЭКО сокращает время до получения бластоцисты хорошего качества и, соответственно, наступления беременности, то есть является методом патогенетического лечения БНГ [5].
Выявленные нарушения раннего эмбриогенеза у пациентов с БНГ обусловили необходимость научного поиска возможных причин и факторов нарушения ранних этапов эмбриогенеза, прежде всего наиболее консервативных – генетических, у пациентов с БНГ.
Ооцит является основным фактором, определяющим эмбриональное развитие человека [25]. Качество ооцита – это способность ооцита успешно оплодотворяться и обеспечивать нормальное развитие эмбриона на ранних стадиях. Существующие в настоящее время сведения не дают основания для заключения, что БНГ вызвано низким качеством ооцитов при удовлетворительном количестве зрелых ооцитов – более 6 на пункцию (в данном исследовании – в среднем 10,8), удовлетворительной частоте оплодотворения (в данном исследовании – 76,5%), наличии возможности криоконсервации эмбрионов в 30% попыток ЭКО (в данном исследовании – 68,1%) и частоте ранних потерь беременности, сопоставимой с общепопуляционной (в данном исследовании – 9,2% в анамнезе) [5, 26]. В то же время невозможно исключить низкое качество ооцитов как причину БНГ [27].
По данным литературы, низкое качество ооцитов при бесплодии может быть обусловлено вариантами генов [11, 12]. Было проведено пилотное исследование – полноэкзомное секвенирование 6 супружеских пар (женщин и мужчин), целенаправленно отобранных из группы БНГ, для того чтобы исключить предполагаемые генетические причины бесплодия у супружеских пар с БНГ.
При полноэкзомном секвенировании наиболее типичных пациентов с БНГ не было обнаружено патогенных и вероятно патогенных вариантов, с которыми может быть связано бесплодие. У женщины из супружеской пары № 1 и мужчины из супружеской пары № 5 были выявлены варианты неясной клинической значимости: FSHR (2-48963163-A-G) и GNRHR (4-67740682-C-T) соответственно, связь которых с БНГ маловероятна. По данным литературы, вариант FSHR (2-48963163-A-G) описан у пациентов с дисгенезией яичников и синдромом гиперстимуляции яичников, однако эти заболевания относились к критериям невключения в группу БНГ. Вариант GNRHR (4-67740682-C-T) описан у пациентов с гипогонадотропным гипогонадизмом, что также было исключено, так как все мужчины из супружеских пар с БНГ имели нормозооспермию на момент включения в исследование и проходили обследование у уролога, по результатам которого данное заболевание не было установлено.
На данном этапе развития науки и фармакологии генетические находки могут повлиять только на решение пациентов об использовании донорских гамет в программах ЭКО; поэтому основная тактика ведения пациентов с БНГ не меняется и заключается в раннем проведении ЭКО с переносом эмбриона хорошего качества (>3, AA, AB, BA по классификации Гарднера) на 5–6-е сутки культивирования [5].
Заключение
Проведено пилотное исследование – полноэкзомное секвенирование 6 супружеских пар (женщин и мужчин), целенаправленно отобранных из группы БНГ на основании клинико-анамнестических данных, лабораторных, инструментальных показателей, результатов использования методов вспомогательной репродукции. У пациентов не было обнаружено известных вариантов, с которыми может быть связано бесплодие.
Необходим дальнейший научный поиск молекулярных механизмов нарушения ранних этапов эмбриогенеза, включающий в первую очередь исследование качества гамет, у пациентов с БНГ.



